


Contreras-González Rosario Angélica, Vázquez-Guzmán Guadalupe Vanessa, Gutiérrez-Chablé Luis Eduardo, Rodríguez-Cardoso Nelly Esther, Blanco-Osorio Nallely, Reyes-Pérez Luis Manuel
Introducción: La vigilancia epidemiológica por laboratorio es uno de los procesos más importantes para la detección de un brote asociado a las infecciones asociadas a la atención de la salud (IAAS), que representan un grave problema de salud pública.
Objetivo: Reconocer la importancia de la identificación bacteriológica en la caracterización de brotes asociados a IAAS.
Materiales y método: Estudio descriptivo, observacional, transversal de noviembre a diciembre 2023 en hospital de segundo nivel, se identificaron patógenos de aislamientos primarios y se realizaron pruebas bioquímicas con perfiles de resistencia de forma manual y automatizada, de brotes asociados a IAAS, cuantificando el tiempo de emisión de resultados.
Resultados: El 100% de aislamientos de Enterobacter cloacae spp los resultados de las pruebas bioquímicas de forma manual se obtuvieron en promedio 46.4±5.1 horas y mediante el equipo automatizado 12.7±3.6 horas, con una diferencia de 33.7 horas, 98-99% de similitud con el microorganismo identificado.
Conclusión:Es importante el empleo de herramientas de identificación bacteriológica automatizadas para la reducción del tiempo en que se puede identificar y controlar un brote.
Introduction: Epidemiological laboratory surveillance is one of the most important processes for detecting an outbreak associated with healthcare-associated infections (HAIs), which represent a serious public health problem.
Objetive: Recognize the importance of bacteriological identification in the characterization of outbreaks associated with HAIs.
Material and methods: Descriptive, observational, cross-sectional study from November to December 2023 in a second-level hospital, pathogens from primary isolates were identified and biochemical tests with resistance profiles were performed manually and automated, of outbreaks associated with HAIs, quantifying the time of issuance of results.
Results: 100% of Enterobacter cloacae spp. The results of the biochemical tests were obtained manually in an average of 46.4±5.1 hours and through automated equipment 12.7±3.6 hours, with a difference of 33.7 hours, 98-99% similarity with the identified microorganism.
Conclusion: The use of automated bacteriological identification tools is important to reduce the time in which an outbreak can be identified and controlled.
Las infecciones asociadas a la atención de la salud (IAAS), anteriormente llamadas infecciones nosocomiales, son aquellas infecciones causadas por gran variedad de hongos, bacterias y virus adquiridos durante la asistencia a la salud en el tratamiento de otras afecciones, ya sea en el período de ingreso en establecimientos de salud o en la atención domiciliaria, esto puede suceder por contaminación cruzada al entrar en contacto con el ambiente de salud, ya que las superficies sirven de abrigo a los microorganismos cuando ocurren fallas en la limpieza ambiental, en el procesamiento de artículos y ropas y/o en el uso de las precauciones-estándar.1 Usualmente, se considera que una infección corresponde a una IAAS si se manifiesta al menos 48 horas después del inicio de la atención médica.2
La aparición de brotes epidémicos en los hospitales asociados a IAAS en la mayoría de los países es un problema en aumento. Este hecho está relacionado con varios factores entre los que destacan: el uso no irracional de antimicrobianos que propician la aparición de microorganismos multirresistentes (especialmente bacterias), deficiencias en la aplicación y supervisión operativa de las precauciones universales y por mecanismos de transmisión, y debilidades en el programa de higiene de manos.3
Así pues, la vigilancia epidemiológica permite monitorear tasas de incidencia de IAAS y brotes asociados de manera intrahospitalaria, como ya se ha mencionado anteriormente y por lo tanto constituye el primer paso indispensable para puntualizar las prioridades locales y nacionales y evaluar la eficacia de las actividades de control de infecciones.4
La vigilancia de las IAAS en México se lleva a cabo mediante la NOM-045-SSA2-2005, Para la vigilancia, prevención y control de las Infecciones Nosocomiales, y el manual de procedimientos estandarizados para la vigilancia epidemiológica. Con el objetivo de generar información de calidad de uso clínico y epidemiológico,5-6 donde se enuncia la vigilancia epidemiológica por laboratorio el cual es uno de los procesos más importantes que permiten la detección de un brote hospitalario asociado a IAAS, es el aislamiento del microorganismo patógeno involucrado, con ello la determinación de su patrón de resistencia antimicrobiana y el análisis de su perfil bioquímico, lo que permite la identificación de un brote. Los tiempos de respuesta a las áreas clínicas en la identificación de patógenos son fundamentales para establecer un tratamiento intencionado en los pacientes, lo que reduce no sólo la morbilidad y mortalidad sino también la propagación de la infección; en el caso del diagnóstico microbiológico existen múltiples variables que pueden afectar la emisión oportuna de resultados, algunas de ellas atribuibles al patógeno y al aislamiento.7
Durante muchos años, la identificación de microorganismos ha dependido de la producción local de medios de cultivos para siembra y realización de pruebas bioquímicas de forma manual, asiladas una de otras por operador dependiente.8 Asimismo, el antibiograma ha sido realizado clásicamente por técnicas de difusión manual que, si bien cada vez se han estandarizado mejor y están en la actualidad adecuadamente validados, es una técnica laboriosa y que requiere de más tiempo para obtener resultados que las técnicas automatizadas disponibles actualmente.9
En cuanto a la identificación bacteriana y los estudios de susceptibilidad, se ha desarrollado un gran número de técnicas rápidas, semiautomatizadas o automatizadas, por lo que estos equipos aportan estandarización y velocidad al proceso.3,10
Los procedimientos convencionales de identificación de microorganismos consisten en observación de características físicas y tintoriales (morfología de colonias y tinción de Gram) y de reacciones bioquímicas.11 Inicialmente, estas pruebas bioquímicas se realizaban en tubos, los que con el tiempo se redujeron en formatos que permiten aumentar el número de pruebas y eventualmente automatizar la lectura de estos resultados.12
Dependiendo de las reacciones de cada microorganismo frente a diferentes sustratos, se obtiene un perfil bioquímico, que al ser comparados con perfiles previamente conocidos permite la identificación del microorganismo.
El tiempo de la identificación convencional era de 24-48 h para permitir la expresión de la reacción, ya que debía lograrse un número crítico de bacterias, que dependían de su velocidad de replicación. Con la automatización se logró reducir este tiempo de 8-10 horas.13
Durante los últimos años, cada vez más laboratorios están implementando la tecnología de espectrometría de masas como el equipo MALDI-TOF que, mediante el análisis proteómico de cepas bacterianas y fúngicas, permite su identificación en minutos. Existen diversos equipos disponibles en el mercado, siendo los más representativos: MALDI-Biotyper de Bruker y Vitek MS, de BioMerieux.14,15
En la actualidad, gracias a este avance tecnológico alcanzado, es posible reducir el tiempo de identificación bacteriológica y a su vez de los brotes hospitalarios para establecer medidas de control y prevención en dichos eventos.16,8 Por ello mediante este trabajo buscamos reconocer la importancia de la identificación bacteriológica en la caracterización de brotes asociados a IAAS mediante los sistemas automatizados.
Para este estudio descriptivo, observacional, prospectivo, transversal, unicéntrico, se llevaron a cabo los procesos de identificación de patógenos de los aislamientos primarios de las muestras biológicas recibidas de noviembre a diciembre del 2023 en el laboratorio de bacteriología clínica del HGZ 20, que tuvieran como resultado el aislamiento del microorganismo Enterobacter cloacae spp, como criterio de inclusión.
La recolección de datos se efectuó bajo la búsqueda intencionada de criterios clínico-epidemiológicos para la asociación de casos relacionados a un brote hospitalario detectado durante ese período. El registro de la información se realizó en un formato de pruebas de susceptibilidad a antimicrobianos, en donde podemos incluir:
Posterior al registro se realizó un análisis de la Identificación bacteriana mediante patrón de susceptibilidad y resistencia antimicrobiana, así como de las reacciones bioquímicas de cada microorganismo aislado de forma manual y de forma automatizada por le Vitek 2 de forma paralela, se incluyeron aquellos casos que compartían variables epidemiológicas (tiempo, lugar y persona), el patrón de susceptibilidad y resistencia, reacciones bioquímicas y el bionúmero.
Los resultados se obtuvieron mediante el cálculo de frecuencias para variables cualitativas. Para variables cuantitativas se utilizaron medidas de tendencia central y desviación estándar.
Se identificaron 3 casos que cumplieron con los criterios para IAAS clasificados como infecciones de torrente sanguíneo, provenientes del área de cunero patológico. En el 100% de cultivos se aisló Enterobacter cloacae spp. Los resultados de las pruebas bioquímicas de forma manual se obtuvieron en promedio con su desviación estándar 46.4±5.1 horas y a la par mediante el equipo automatizado obteniendo un perfil bioquímico reportado por el Vitek 2, de 12.7±3.6 horas, la diferencia entre ambos procesos fue de 33.7 horas, con 98-99% de similitud con el microorganismo identificado, con un nivel de confianza reportado como: Identificación excelente. Ver cuadros I, II y III.
CUADRO I. Patrón de susceptibilidad y resistencia antimicrobiana y perfil bioquímico del paciente.1
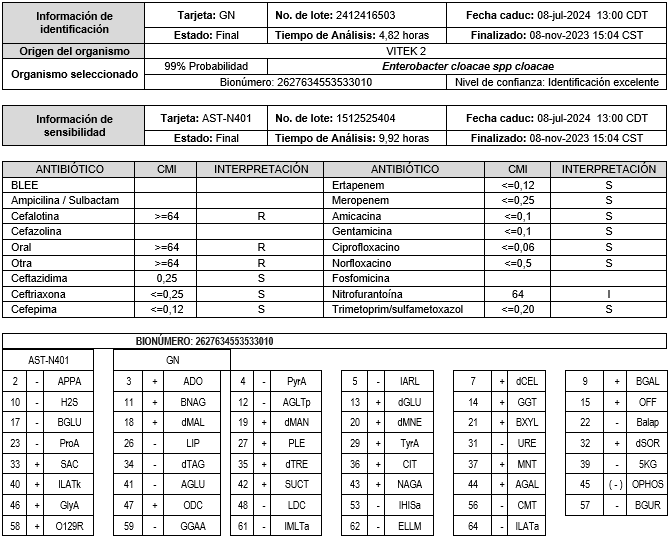
CUADRO II. Patrón de susceptibilidad y resistencia antimicrobiana y perfil bioquímico del paciente.2
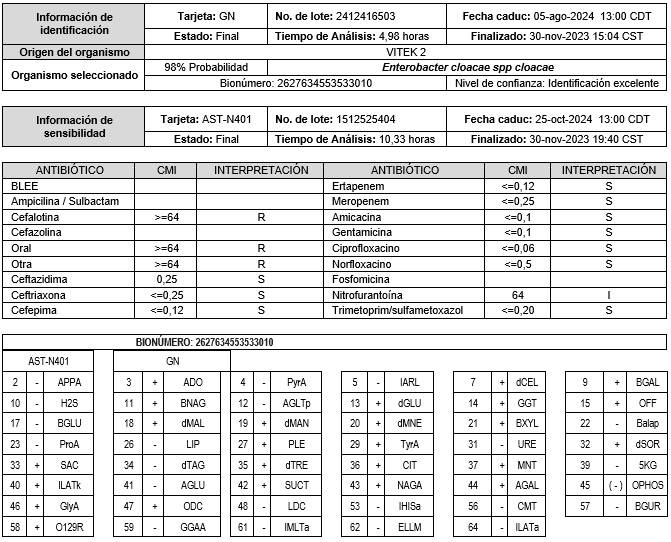
CUADRO III. Patrón de susceptibilidad y resistencia antimicrobiana y perfil bioquímico del paciente.3
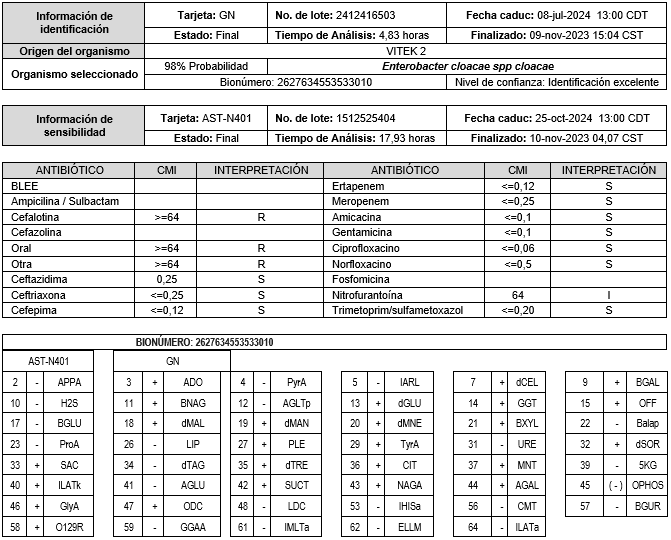
La identificación rápida y precisa de microorganismos patógenos es importante en el manejo de las IAAS, especialmente en el contexto de brotes epidémicos en hospitales. En estudios previos que han documentado la creciente prevalencia de Enterobacter cloacae spp en entornos hospitalarios, lo que subraya la importancia de la vigilancia epidemiológica en la detección temprana de brotes.8, 9
Los resultados obtenidos muestran una clara ventaja en el uso de sistemas automatizados en comparación con los métodos manuales. La reducción en el tiempo de respuesta es fundamental, ya que permite implementar medidas de control y tratamiento más rápidamente, disminuyendo así la morbilidad y mortalidad asociadas a estas infecciones. Un estudio realizado por Johnson y Patel10 encontró que la implementación de sistemas automatizados en laboratorios de microbiología permitió una reducción del 50% en el tiempo de identificación de patógenos, lo que se tradujo en una disminución significativa de las tasas de IAAS en un entorno hospitalario. Kumar y Singh11 también corroboran estos hallazgos, indicando que los sistemas automatizados pueden reducir el tiempo de identificación en un 60%, permitiendo un tratamiento más oportuno.
La rapidez en la identificación de patógenos es esencial para establecer un tratamiento dirigido, lo que puede ser determinante en el pronóstico de los pacientes. Además, la capacidad de identificar brotes de manera oportuna permite a los hospitales aplicar medidas de control más efectivas, como la mejora en la higiene de manos y la aplicación adecuada de precauciones universales. Según un estudio de Lee y Kim12, la identificación temprana de microorganismos multirresistentes resultó en una reducción del 30% en la mortalidad asociada a infecciones nosocomiales. Martínez y Torres13 documentaron que la implementación de tecnologías de identificación rápida en un hospital universitario resultó en una reducción del 40% en la duración de las infecciones nosocomiales, destacando la importancia de la tecnología en el control de brotes.
Aunque los resultados son prometedores, es importante considerar algunas limitaciones. La muestra de casos analizada es pequeña, lo que puede limitar la generalización de los hallazgos. Futuros estudios deberían incluir un mayor número de casos y explorar la efectividad de estos sistemas automatizados en diferentes tipos de infecciones y en diversos entornos hospitalarios. Investigaciones adicionales, como la de Nguyen y Tran14, que se centró en la identificación de patógenos multirresistentes, demuestran que el uso de sistemas automatizados no solo acelera el proceso de identificación, sino que también mejora la precisión en la detección de cepas resistentes, lo cual es crucial para el manejo adecuado de las infecciones. Por tanto, la implementación de tecnologías automatizadas para la identificación de microorganismos representa un avance significativo en la lucha contra las IAAS. Este estudio resalta la necesidad de continuar invirtiendo en estas tecnologías para mejorar la respuesta frente a brotes epidémicos y garantizar una atención médica de calidad.
Los resultados de este estudio reconocen la importancia que tiene la reducción del tiempo en la identificación microbiológica precisa de los agentes causales involucrados en brotes IAAS en entornos hospitalarios, mediante herramientas de identificación bacteriológica automatizadas, como el equipo Vitek 2.

Contreras-González Rosario Angélica
Hospital General de Zona No. 20, IMSS Puebla.
Vázquez-Guzmán Guadalupe Vanessa
Hospital General de Zona No. 20, IMSS Puebla.
Gutiérrez-Chablé Luis Eduardo
Hospital General de Zona No. 20, IMSS Puebla.
Rodríguez-Cardoso Nelly Esther
Hospital General Regional No. 36 “San Alejandro”, IMSS Puebla.
Blanco-Osorio Nallely
Hospital General de Zona No. 20, IMSS Puebla.
Reyes-Pérez Luis Manuel
Unidad de Medicina Familiar No. 57, IMSS Puebla.
CONTACTO
Contreras-González Rosario Angélica
Calle Encino #352 colonia El Cerrito, C.P. 72440, Heroica Puebla de Zaragoza, Puebla
E-mail: angycg92@gmail.com
RECIBIDO: 7 de abril de 2025
ACEPTADO: 20 de mayo de 2025
Introducción del autor